Một trong những cách quan trọng để cải thiện khả năng chịu mặn của lúa là xác định các gen chịu mặn để tạo ra các giống mới chống chịu mặn.
Ảnh hưởng của căng thẳng phi sinh học chịu trách nhiệm cho sự suy giảm ước tính 50-70% trong sản xuất cây trồng trên toàn thế giới. Trong số các căng thẳng phi sinh học này, độ mặn được coi là một trong những hạn chế môi trường chính đối với năng suất nông nghiệp. Khoảng 20% diện tích đất canh tác bị ảnh hưởng (~ 45 triệu ha) do độ mặn tăng trên toàn thế giới (Gupta, và ctv.,2014). Cây lúa là cây ngũ cốc quan trọng nhất nhưng rất nhạy cảm với muối. Độ mẫn cảm với độ mặn thay đổi ở các giai đoạn khác nhau trong thời kỳ tăng trưởng của nó. Cây lúa mẫn cảm nhất ở giai đoạn cây con và giai đoạn sinh sản (Lang và ctv.,2020) nhưng tương đối chịu được ở giai đoạn nảy mầm (Lang và ctv.,2017). Hơn nữa, khả năng chịu đựng của cây con và giai đoạn sinh sản rất kém, cho thấy các bộ gen riêng biệt có thể tham gia vào các giai đoạn phát triển khác nhau trong việc khắc phục căng thẳng muối (Moradi và ctv.,2007). Có một số nghiên cứu biểu hiện gen giai đoạn cây con ở lúa nhưng rất ít (Walia và ctv.,2007) ở giai đoạn sinh sản. Ở giai đoạn sinh sản, khả năng sinh sản và số lượng bông trên mỗi cây được báo cáo là thành phần năng suất bị ảnh hưởng nhiều nhất dưới áp lực muối (Kawasaki, và ctv.,2001). Hiểu rõ hơn về cơ chế chịu mặn ở lúa ở giai đoạn sinh sản là một bước cần thiết trong nỗ lực cải thiện hạt dưới áp lực muối đặc biệt là chọn lọc trong quần thể lai.
 |
| "Mặn là một yếu tố môi trường quan trọng ảnh hưởng đến sự tăng trưởng và phát triển của cây lúa"- (GS.TS Nguyễn Thị Lang) |
Vật liệu và phương pháp
Vật liệu: hai tổ hợp lai OM10252/Pokkali//OM10252 và OM10252/OM4900//OM10252.
Thí nghiệm thanh lọc mặn trong dung dịch dinh dưỡng Yoshida (theo phương pháp IRRI, 1997); Lang và ctv 2015. Phân tích kiểu gen theo Lang. 2016. Phân tích bản đồ gen theo Lang và ctv 2019.
Kết quả và thảo luận
Thiết lập liên kết gen mặn bằng chi thị phân tử SSR
Thiết lập liên kết gen dựa vào bản đồ liên kết gen trên cơ sở chỉ thị phân tử được thực hiện với quần thể BC2F2 của cặp lai OM10252/Pokkali//OM10252. Đối với SSR hay microsatellite phản ánh thể đa hình trên cơ sở số đơn vị lặp lại của một vùng mục tiêu mang gen mặn của genome cây lúa. Kết quả sản phẩm đa hình để thiết lập bản đồ di truyền trên lúa hơn 230 SSR marker, khoảng cách trung bình là 10,91 cM. Số lượng và thành phần của microsatellite thay đổi trên các cặp lai khác nhau. Tần suất lặp lại chuỗi mã dài hơn 20 bp. Chuỗi mã nucleotide kế cận chuỗi mã lặp lại được dùng để thiết kế primer nhằm khuếch đại số lượng khác nhau của đơn vị lặp lại trong những giống khác nhau. Bản đồ di truyền trên quần thể BC2F2 của tổ hợp OM10252/Pokkali được ghi nhận (Lang và ctv.,2019). Với 230 chỉ thị nằm trên 12 nhiễm sắc thể.
Phân tích QTL mặn
Hầu hết các tính trạng nông học quan trọng như năng suất, yếu tố hình thành năng suất, ngày trổ hoa, khô hạn v.v… được kiểm soát bởi đa gen. Hoạt động đa gen và sự tương tác lẫn nhau, hiện vẫn còn được biết rất ít trong khi chúng điều khiển tính trạng số lượng. Các yếu tố cải tiến tính trạng số lượng (QTMF = quantitative trait modifying factor) cũng được xác định như những loci ảnh hưởng đến tính trạng số lượng thông qua epistasis. Trong khi đó, QTL có ảnh hưởng kiểu hình tương đối lớn và độc lập trên cơ sở di truyền. Marker phân tử có liên kết chặt với gen điều khiển gen mặn trên tính trạng mặn được đánh giá. Đối với cây lúa các tính trạng mặn trên nhiều chỉ tiêu. Tuy nhiên trong đề tài này tập trung vào 4 tính trạng được phân tích, đã được áp dụng QTL marker để phân tích. Bản đồ liên kết gen với mật độ dày cũng đã được thiết lập với sự trợ giúp của phần mềm MAPMARKER/QTL.
Kết quả tương tự cũng đã được ghi nhận trong một nghiên cứu gần đây sử dụng dòng hồi giao IR28/Pokkali, trong đó dòng không Saltol đạt mức trung bình điểm SES là 6,3 so với IR29 ở SES = 9 với điều kiện stress mặn của 18 dSm-1 (Lang et al., 2017). Đó là nghiên cứu sử dụng phân tích marker duy nhất trên 65 marker SSR với 181 cá thể BC3F4 để lập bản đồ QTL cho SES trên nhiễm sắc thể 3, 5, 6, 10, và 11, mặc dù trong các vùng khác nhau hơn nghiên cứu này, ngoại trừ một QTL thông thường trên nhiễm sắc thể số 3 tại RM3867. Phát triển NIL thêm QTLs Pokkali có nguồn gốc là cần thiết để nghiên cứu cơ chế chống chịu mặn được điều khiển bởi các QTLs bổ sung và nếu chúng hoạt động trong một phương thức bổ sung kết hợp với Saltol trên quần thể nầy OM10252/Pokkali.
Thông thường người ta rất khó xác định vị trí chính xác và hoạt động của các loci tính trạng số lượng. Để khắc phục nhược điểm trên, chúng tôi thành lập các nguồn vật liệu di truyền (genetic stocks) có tính trạng được biết rõ ràng, thí dụ như các dòng gần đẳng gen (NIL), mang một hoặc nhiều đoạn nhiễm sắc thể của một dòng bố mẹ, trên nền tảng di truyền của một dòng bố mẹ khác. Thực hiện công tác điều tra trên toàn genome để xác định đoạn nhiễm sắc thể được du nhập vào bằng phương tiện SSR. Nhờ sử dụng các dòng NIL đối với một đoạn nhiễm sắc thể nào đó, có thể xử lý một QTL nào đó như một yếu tố đơn trong di truyền Mendel. Bản đồ SSR có chất lượng cao, có mật độ dày các marker, sẽ giúp cho công tác xác định dễ dàng hơn. Nó còn giúp cho ta lập được bản đồ và xử lý các tính trạng số lượng thành các yếu tố di truyền Mendel. Phương pháp này bao gồm: (i) tìm kiếm sự kết hợp giữa những SSR marker tạo dòng đang phân ly, và (ii) xác định sự ích lợi của một quần thể phân ly để tìm thấy liên kết của marker với QTL.
Trên quần thể bản đồ di truyền trên quần thể BC2F2 của tổ hợp OM10252/Pokkali với 250 cá thể phủ trên 223 marker trên 12 nhiễm sắc thể. Chỉ có 3 QTL nằm trên nhiễm sắc thể được đánh giá tập trung trên 3 nhiễm sắc thể: 1, 3 và số 9.
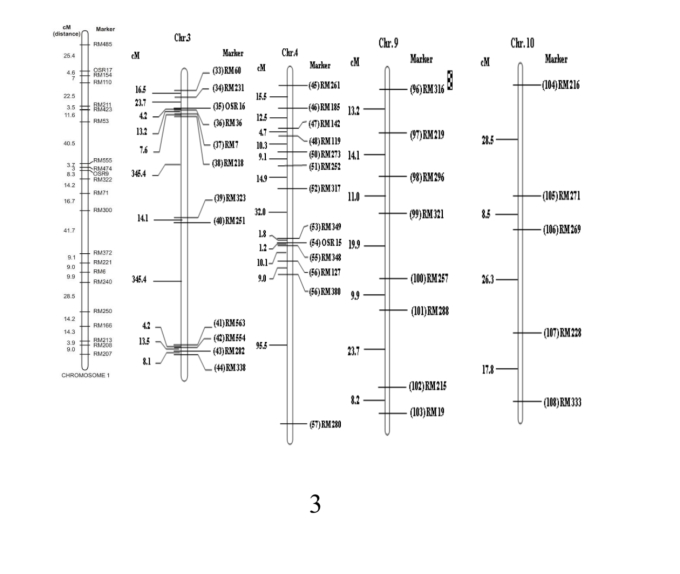 |
| Hình 1: Bản đồ trên ba nhiễm sắc thể 1, 3 và 9 trên quần thể OM10252/Pokkali//OM10252 |
Phát triển dòng đẳng gen (NIL) thêm phân tích QTLs cho thấy giống Pokkali có nguồn gốc là cần thiết để nghiên cứu cơ chế chống chịu mặn được điều khiển bởi các QTLs bổ sung và nếu chúng hoạt động trong một phương thức bổ sung kết hợp với Saltol trên quần thể con lai của OM10252/Pokkali//OM10252.
Phát triển hệ thống MAB tối ưu hóa cao cho sự du nhập gen chịu mặn
Trong vụ Đông Xuân 2020 tiến hành thu nhận hạt F1 được tạo ra trong vụ trước, gieo và tạo hạt hồi giao lần thứ nhất (BC1). Các con lai F1 đều thuần nhất về tính kháng, do đó trên mỗi quần thể lai đã chọn 30 cây để làm mẹ, lấy phấn bố trên các giống OM4900 và tương ứng để tạo hạt hồi giao, mỗi cây tiến hành lai từ 5 – 7 hạt. Kết quả đã tạo được cây hồi giao với giống OM 10252. Bên cạnh đó, trên các giống tái tục và giống cho gen kháng tiếp tục tiến hành tự thụ được từ 20-30 cây cung cấp cho các thí nghiệm tiếp theo.
Kết quả đánh giá sàng lọc cây mang gen kháng mặn từ các quần thể mang gen mặn
Ứng dụng 3 chỉ thị phân tử RM223, RM3252-S1-1, HATRI 2 để đánh giá gen kháng chịu mặn trên 30 cá thể của quần thể BC2 từ OM10252/OM4900//OM10252. Dùng các chỉ thị phân tử liên kết với các dòng lai của bốn tổ hợp với 3 đoạn mồi SSR để tạo sự khuếch đại chuyên biệt cho phân tích trên gen khô hạn là chỉ thị phân tử RM223 và RM3252-S1-1, đây là ba chỉ thị phân tử chuyên biệt có liên quan đến mặn được công bố vào năm 2007 (Lang và ctv, 2007) và HATRI 2 (Lang, 2017). Các chỉ thị phân tử này được ghi nhận kết quả là tất cả sản phẩm của SSR đều cho sự đa hình trên 30 dòng lúa (P=100%).
Kết quả lai tạo quần thể hồi giao OM10252/OM4900//OM10252
Quần thể OM10252/OM4900//OM10252 bắt đầu được lai tạo từ năm 2016 và các cá thể con lai liên tục được chọn lọc dựa trên chỉ thị phân tử cho đến thế hệ BC3, các cá thể ở mỗi thế hệ được lựa chọn dựa trên gen dị hợp tử mặn từ bố và đồng hợp tử ở 3 chỉ thị phân tử (RM223, RM3252-S-1-1, HATRI 2) cho các gen đánh dấu trên cá thể mẹ.
Khuếch đại DNA bằng phương pháp PCR - SSR với marker RM223
Gen mục tiêu được chọn để thực hiện thí nghiệm này là gen kháng mặn trên nhiễm sắc thể số 8 (saltol). Gen liên kết chặt trên nhiễm sắc thể số 8 được đánh dấu bởi marker phân tử RM223. Gen nàycó liên kết gen chống chịu mặn ở giai đọan mạ và phát dục (Nguyễn Thị Lang và ctv, 2001). Marker RM223 được sử dụng làm marker đánh dấu có kích thước là 200-220 bp và được dùng làm khuôn DNA để thiết lập các cặp primer đặc hiệu. Kết quả ghi nhận trên hình 2 cho thấy đa số các cột đều có dạng dị hợp, xuất hiện với hai băng có kích thước 200 bp – 220 bp tương ứng với bố mẹ cho gen kháng mặn và không mặn.
Trên quần thể OM10252/OM4900 ghi nhận vị trí P1 tương ứng với DNA của OM10252 và P2 tương ứng với OM4900 ghi nhận với RM223 cho thấy vị trí DNA của OM10252 là 220bp và kích thước phân tử của OM4900 là 200bp. Vị trí con lai BC2F1 mang cả gen bố và mẹ. Chỉ trừ dòng số 5 cho alen giống kích thước của mẹ và dòng số 7 cho alen giống kích thước của bố là OM4900. Chứng tỏ rằng trên quần thể này RM223 cho đa hình. Như vậy quần thể này cho thấy gen của bố và mẹ đã chuyển sang con lai. Do đó tiếp tục trồng cho lai tiếp thế hệ BC3F1.
 |
| Hình 2: Sản phẩm PCR của các dòng tại locus RM223, liên kết với gen kháng mặn tại nhiễm sắc thể số 8 vị trí hai băng 220bp và 200bp trên gel agarose 3%. Ghi chú: M: là marker chuẩn; P1: OM10252, P2: OM4900, 1-30 là cây lai BC2F1 |
Tương tự phân tích kiểu alen của chỉ thị RM3252-S-1-1 do Viện lúa Quốc tế thiết kế cho gen chống chịu mặn. Ghi nhận kích thước phân tử cho vị trí của Pokkali trên chỉ thị RM3252-S-1-1 này có kích thước là 220bp. Điều này cũng phù hợp với Mohammadi và ctv (2010). Kết quả ghi nhận đa số các giống khác biệt với giống OM4900.
Quần thể OM10252/OM4900 ghi nhận vị trí P1 tương ướng với DNA của OM10252 và P2 tương ứng với OM4900 ghi nhận với RM3252-S1-1 cho thấy vị trí DNA của OM10252 là 230bp và kích thước phân tử của OM4900 là 220bp. Vị Trí con lai BC2F1 mang cả bố và mẹ. Chỉ trừ một dòng 1, cho alen giống kích thước của bố. Chứng tỏ rằng trên quần thể nầy RM3252-S1-1 cho đa hình. Như vậy quần thể này cho thấy gen của bố mẹ đã chuyển sang con lai. Từ kết quả trên tiếp tục trồng BC2F1 để cho lai tiếp thế hệ BC3F1.
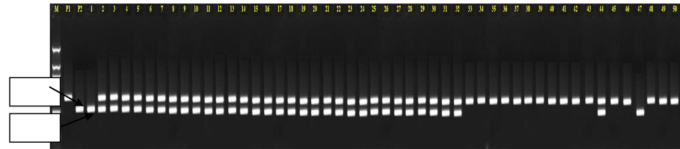 |
| Hình 3: Sản phẩm PCR của các dòng tại locus RM3252-S1-1, liên kết với gen kháng mặn tại nhiễm sắc thể số 1 vị trí hai băng 230bp và 220bp trên gel agarose 3% Ghi chú: M: là marker chuẩn;P1:OM10252, P2:OM4900, 1-30 là cây lai BC2F1 |
Đối với tổ hợp OM10252/OM4900//OM10252 trên chỉ thị HATRI 2
Trong quần thể OM10252/OM4900 ghi nhận vị trí P1 tương ướng với DNA của OM10252 và P2 tương ứng với OM4900 ghi nhận với HATRI 2 cho thấy vị trí DNA của OM10252 là 300bp và kích thước phân tử của OM4900 là 250bp. Vị trí con lai F1 mang cả gen bố và mẹ. Mười dòng BC1F1 số 1, 14, 15, 16, 24, 25, 26, 27, 28 và số 30 cho alen giống kích thước của mẹ. Một dòng 29 có kích thước giống với bố. Chứng tỏ rằng trên quần thể nầy HATRI 2 cho đa hình. Như vậy quần thể này cho thấy gen của bố mẹ đã chuyển sang con lai. Từ kết quả trên tiếp tục trồng BC2F1 để cho lai tiếp thế hệ BC3F1.
M P1 P2 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30
 |
| Hình 4: Sản phẩm PCR của các dòng tại locus HATRI 2, liên kết với gen kháng mặn tại nhiễm sắc thể số 1 vị trí hai băng 300bp và 250bp trên gel agarose 3% Ghi chú: M: là marker chuẩn; P1: OM 10252; P2: OM 4900; 1-30 là cây lai BC2F1 |
Sau khi thanh lọc mặn bằng 3 chỉ thị RM223, HATRI 2 và RM3252-S-1-1, tiến hành chọn lọc 10 dòng số 1, 14, 15, 16, 24, 25, 26, 27, 28, 30 để đánh giá hàm lượng amylose. Đánh giá kiểu gen trên chỉ thị phân tử Wx nằm trên nhiễm sắc thể số 6. Với chỉ thị này cho sản phẩm khuếch đại DNA là 100%. Sản phẩm khuếch đại cho đa hình với hai kích thước phân tử khác nhau là 200bp với 220bp trên 10 dòng chọn lọc bằng chỉ thị phân tử.
Tám dòng BC3F2 được cho tự thụ tiếp tục thành BC3F3 để trồng và đánh giá năng suất. Kết quả ghi nhận có ba dòng cho năng suất vượt trội so với bố mẹ đó là các dòng số 1 (BC3F3-1), số 2 (BC3F3-2) và số 8 (BC3F3-48). Trong đó dòng số 2 (BC3F3-2) và dòng số 8 (BC3F3-48) cho năng suất bằng nhau (7,9 tấn/ha). Tuy nhiên về khả năng chống chịu độ mặn thì dòng số 2 chống chịu độ mặn không cao.
Đánh giá các dòng triển vọng trong vụ Hè Thu 2022 trên cơ sở năng suất và thành phần năng suất đã chọn được dòng BC3F4-48 triển vọng và được đặt tên là HATRI 174.
Kiểm tra các dòng mang gen mặn trong nhà lưới
Thanh lọc các dòng trong nhà lưới mang gen mặn. Có 11 dòng đưa ra trồng tiếp tục để đánh giá mặn trện các giai đoạn mạ. Kết quả trong giai đoạn nầy có các dòng BC3F4-2; BC3F4-42; BC3F4-48 chịu tốt giai đoạn mạ (Bảng2).
Thanh lọc các dòng phân ly ở các giai đoạn: giai đoạn đẻ nhánh và giai đoạn trổ.
Dựa trên các tính trạng trên chọn lọc các dòng để đánh giá và thanh lọc. Kết quả các dòng cho năng suất cao để thanh lọc mặn với 4 chỉ tiêu liên quan. Trọng lượng khô của rễ, trọng lượng khô của thân, chiều dài của rễ, chiều cao cây.
Kết quả ghi nhận: Qua chọn lọc quần thể OM10252/OM4900//OM10252 có tám dòng (BC3F4-1, BC3F4-2, BC3F4-6, BC3F4-24, BC3F4-30, BC3F3-36, BC3F4-42, BC3F4-48) mang cả ba gen: chịu mặn, năng suất cao và hàm lượng amylose thấp trên 4 chỉ thị RM3252-S1-1, HATRI 2, Wx và RM223. Đã chọn được dòng BC3F4-48 có năng suất cao, độ thuần tốt nhất đặt tên là HATRI808. Dòng này sẽ được đánh giá và chọn lọc các thế hệ tiếp theo để đánh giá về độ thuần các tính trạng khác.
Kết luận
Trên quần thể bản đồ di truyền trên quần thể BC2F2 của tổ hợp OM10252/Pokkali với 250 cá thể phủ trên 223 marker trên 12 nhiễm sắc thể. Chỉ có 3 QTL nằm trên nhiễm sắc thể được đánh giá tập trung trên 3 nhiễm sắc thể: 1, 3 và số 9 mang gen mặn được thiết lập.
Ứng dụng trong chọn lọc quần thể OM10252/OM 4900//OM 10252 có tám dòng (BC3F4-1, BC3F4-2, BC3F4-6, BC3F4-24, BC3F4-30, BC3F3-36, BC3F4-42, BC3F4-48) mang cả ba gen: chịu mặn, năng suất cao và hàm lượng amylose thấp trên 4 chỉ thị RM3252-S1-1, HATRI 2, Wx và RM223. Đã chọn được dòng BC3F4-48 có năng suất cao, độ thuần tốt nhất đặt tên là HATRI808. Dòng này sẽ được đánh giá và chọn lọc các thế hệ tiếp theo để đánh giá về độ thuần các tính trạng khác.
Nhóm tác giả Viện Nghiên Cứu Nông Nghiệp Công Nghệ Cao ĐBSCL, Cần Thơ (HATRI))


















Hội nghị Nữ khoa học toàn quốc lần thứ III - 2023: Nâng cao vai trò của Nữ trí thức trong nghiên cứu khoa học và ứng dụng KHCN
Hội nghị Nữ khoa học toàn quốc lần thứ III 2023: " “Ứng dụng KHCN vào đời sống, xã hội và chăm sóc sức khỏe cộng đồng”
60 báo cáo khoa học tham gia Hội nghị Nữ khoa học lần thứ III-2023
Hội Nữ Trí thức Việt Nam tổ chức thành công Hội nghị Nữ khoa học toàn quốc lần thứ II
Nhà khoa học nữ giành Nobel kinh tế 2023
Chân dung khoa học nữ đạt giải Nobel Vật lý 2023 nắm giữ kỷ lục tạo xung laser
Cùng chuyên mục
Bất ngờ loài cá sở hữu trí thông minh của động vật có vú, có khả năng tự nhận thức
Phát hiện di tích pháo đài cổ 1000 năm còn nguyên vẹn giữa rừng sâu Ba Lan
Hổ phách 99 triệu năm lần đầu hé lộ chứng u xương thời cổ đại
Giải mã hóa thạch “quái thú” cổ đại giữa sa mạc Sahara
Trung Quốc phát triển mô hình AI kết nối dữ liệu thiên văn toàn cầu
Nghiên cứu mới viết lại thời điểm cuộc di cư sớm nhất của loài người